生物体内的各项生命进程均呈现出动态演进的特性,涵盖了细胞自发育、分化至对外部刺激响应的各个阶段,其间伴随着基因表达的连续性变化。得益于单细胞测序技术的革新,科研人员现今能够实时捕获并深入解析细胞在不同时间节点的内在状态与行为特征,从而在分子水平上实现对细胞动态变化轨迹的精准追踪。这一技术突破为生物医学研究领域带来了颠覆性的变革,使得多阶段scRNA-seq数据分析成为当前研究实践中的主流手段。
在进行scRNA-seq数据的多阶段分析时,研究者通常会着重关注基因表达水平随时间推移或生物进程推进所发生的显著变化,这些被称为多阶段动态差异基因,它们很可能在对应的生物过程中扮演关键角色。近年来,学术界涌现出一系列专门针对scRNA-seq数据动态差异表达基因识别的方法与工具,如tradeSeq和Monocle2等。这些方法的核心在于如何有效利用在一定阶段间隔内采集的数据,重构出单个细胞基因表达随时间动态演进的过程。
然而,随着单细胞测序实验在包含多种表型乃至跨物种、人群样本中的广泛应用,数据集的多样性日益增加,导致细胞类型的跨样本差异性成为单细胞分析中一个新的复杂维度。对于分析人员而言,除了要应对细胞固有的异质性外,还需充分考量样本间异质性这一额外层次的影响,以确保对复杂生物过程的精准刻画与深度理解。换言之,科研人员在处理此类数据时,必须同时兼顾细胞个体特异性与样本间群体差异,才能确保对生物体系动态变化的全面、准确揭示。

近日,西安交通大学公共卫生学院孙世权教授团队在《基因组生物学》(Genome Biology,IF=12.30)上发表了《用TDEseq高效准确检测多样本多阶段单细胞转录组学数据中的时间基因表达模式》(Powerful and accurate detection of temporal gene expression patterns from multi-sample multi-stage single-cell transcriptomics data with TDEseq),作者开发了一种高效灵活的非参数方法,用于检测多个时间点上的基因表达模式。方法称为TDEseq,即时间序列scRNA-seq数据的时间差异表达基因。
TDEseq采用线性可加混合模型(LAMM)来拟合单个基因表达值和时间点的关系,通过引入形状约束的样条函数表征基因表达水平的时间序列动态变化过程检测具有特定表达模式的时间动态差异基因,引入随机效应项以控制样本间的异质性,并最终产生统计学上严格的p值。TDEseq不仅保证了更高的检验效力(power),也实现了更好的对错误发现率的控制(FDR control),尤其是在处理样本异质性强的多样本scRNA-seq数据时。在这个模型中,通常引入二次I样条和三次C样条作为基函数,这有助于检测四种潜在的时间基因表达模式,即生长、衰退、高峰或低谷。最终,通过广泛的模拟研究,发现TDEseq可以在整个转录组水平上正确控制类型I错误率,并在功率模拟中显示出检测时间表达基因的强大性能。
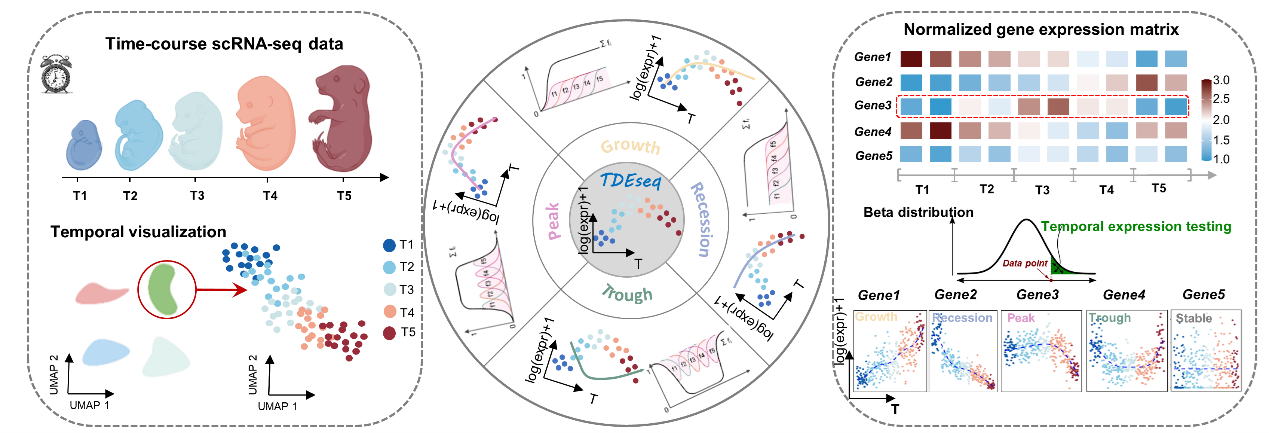
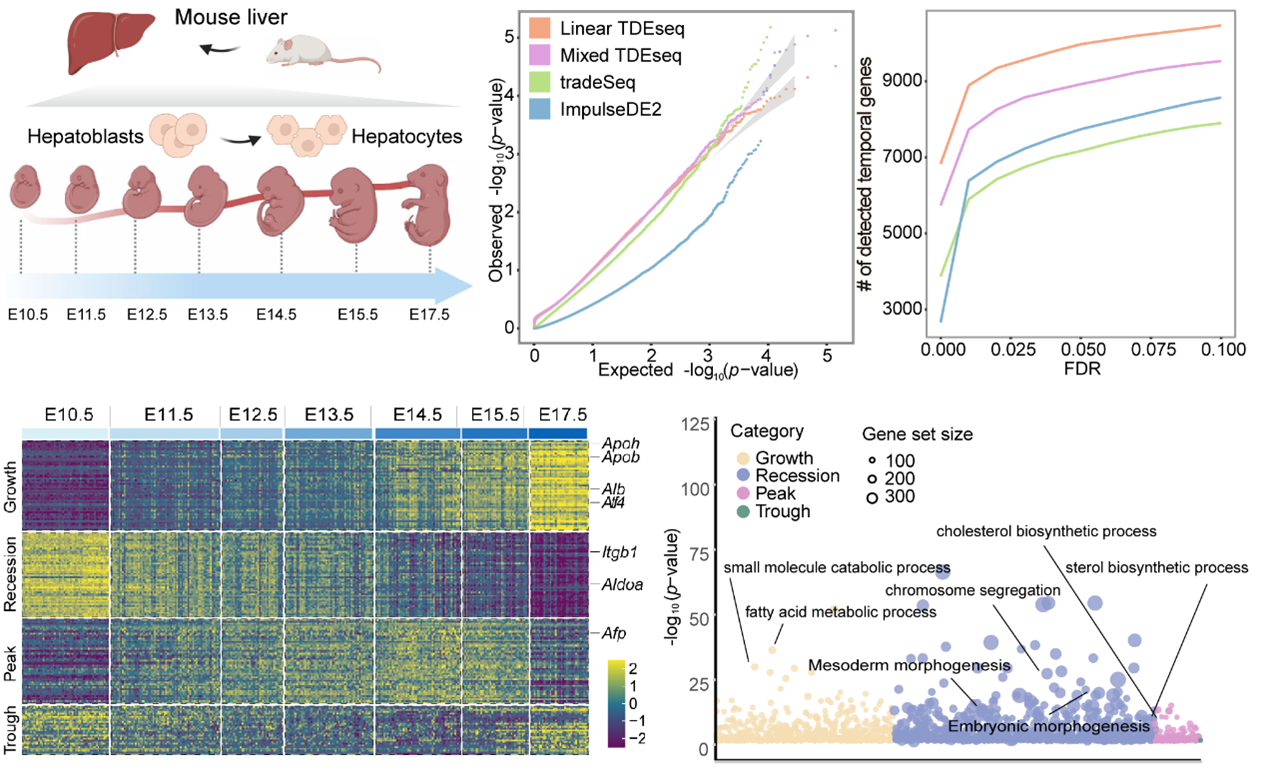
TDEseq在癌细胞系药物响应、小鼠肝脏胚胎发育、肺腺癌进展过程、NK细胞相应SARS-CoV-2病毒感染等时间序列scRNA-seq数据中均展现出良好的性能。以小鼠肝脏胚胎发育为例,TDEseq识别的动态差异表达基因比tradeSeq多20%。TDEseq所识别的动态差异表达基因不仅具有明确的时间动态表达模式,且与肝脏胚胎发育过程呈强相关性。
西安交通大学助理研究员樊越为该论文的唯一第一作者;西安交通大学孙世权教授为该论文的独立通讯作者。公共卫生学院单细胞组学与健康研究中心、国家卫健委环境与地方性疾病重点实验室、环境与疾病相关基因教育部重点实验室、陕西省疾病防控与健康促进重点实验室为论文通讯作者单位。该研究成果受到了国家自然科学基金优秀青年基金项目、科技部重点研发计划项目、国家自然科学基金项目、西安交通大学青年拔尖人才项目等资助。
TDEseq的R语言软件包链接:https://sqsun.github.io/software.html